Apache Liminal : quand MLOps rencontre GitOps

Apache liminal est un logiciel open-source qui propose une solution pour déployer des pipelines de Machine Learning de bout en bout. En effet, il permet de centraliser toutes les étapes nécessaires à la construction de modèles de Machine Learning, du nettoyage des données au déploiement des modèles.
Cette solution propose une approche déclarative pour les projets MLOps. Le pipeline qui encapsule les différentes étapes de préparation, de formation et de déploiement de votre Machine Learning est écrit en YAML.
Ce fichier, et les scripts Python vers lesquels il pointe, sont facilement versionnés à l’aide d’outils comme Git, ouvrant la porte à une pratique GitOps. GitOps décrit une architecture dans laquelle le système est reproductible à partir de l’état stocké dans un référentiel Git. Les ingénieurs de données et les data scientists peuvent alors collaborer ensemble pour améliorer le modèle.
Apache Liminal exploite Flux d’air Apache, Docker et Kubernetes afin de créer et de déployer notre pipeline.
Installation
Pour reproduire toutes les commandes trouvées dans cet article, Apache Liminal nécessite d’installer Docker et Kubernetes sur votre machine. Kubernetes peut être installé avec minikube.
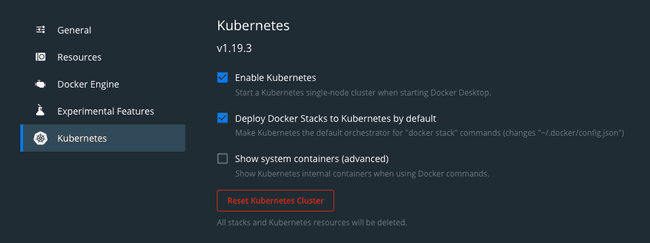
Si vous êtes sur MacOS avec Docker déjà installé, l’approche la plus simple consiste à activer Kubernetes en cochant la case intitulée “Déployer les piles Docker sur Kubernetes par défaut” dans Docker Desktop.
Ensuite, vous pouvez installer Apache Liminal en utilisant pip.
pip install
git+https://github.com/apache/incubator-liminal.gitCréation d’un pipeline Liminal
Création des scripts Python
Commençons par créer un dossier à la racine de notre répertoire de projet pour rassembler tous les scripts Python nécessaires à notre pipeline.
À l’intérieur, nous créons d’abord notre requirements.txt fichier pour la gestion des dépendances. Apache Liminal utilisera ce fichier pour installer tous les packages Python répertoriés nécessaires pour assurer le bon fonctionnement de nos scripts sur les images Docker. Dans notre exemple, nous allons utiliser les packages suivants :
urllib3
pandas
numpy
tensorflow
scikit-learnDans notre cas d’utilisation, l’étape de préparation des données sera principalement réduite au téléchargement du jeu de données. Nous allons utiliser le qualité-du-vin.csv fichier pour former notre modèle. Comme nous le verrons plus loin, ces données seront directement accessibles depuis les pods.
Nous allons créer un fichier nommé download.py qui contiendra toute la logique pour télécharger le fichier et nettoyer les données :
import urllib3
import pandas as pd
import numpy as np
import os
PATH = "/mnt/data/"
file_path = str(PATH) + "file.csv"
http = urllib3.PoolManager()
url = os.environ['url']
r = http.request('GET', url)
if os.path.exists(file_path):
os.remove(file_path)
else:
print("file not exist")
with open(file_path, 'xb') as f:
f.write(r.data)
dataset = pd.read_csv(file_path)
for field in dataset.columns:
if type(dataset[field][0]) == np.int64 :
new_field = field.replace(' ', '_')
dataset = dataset.rename(columns=field : new_field)
print('i - field = ' + str(new_field))
elif type(dataset[field][0]) == np.float64 :
new_field = field.replace(' ', '_')
dataset = dataset.rename(columns=field : new_field)
print('f - field = ' + str(new_field))
dataset.to_csv(file_path, index=False)Ici, nous obtenons le fichier en utilisant une variable d’environnement nommée url qui est défini dans notre script YAML comme suit :
env_vars:
url: "https://raw.githubusercontent.com/mlflow/mlflow/master/examples/sklearn_elasticnet_wine/wine-quality.csv"Ensuite, nous créons un script python nommé wine_linear_regression.py pour former notre modèle :
import os
import sys
import numpy as np
import pandas as pd
import tensorflow as tf
from six.moves import urllib
from sklearn.model_selection import train_test_split
PATH = "/mnt/data/"
path = str(PATH) + "file.csv"
dataset = pd.read_csv(path)
labels = dataset['quality'].tolist()
dataset = dataset.drop(["quality"], axis=1)
x_train, x_test, y_train, y_test = train_test_split(dataset,
labels,
train_size=0.9)
NUMERIC_COLUMNS = ['alcohol', 'chlorides', 'citric_acid', 'density', 'fixed_acidity',
'free_sulfur_dioxide', 'pH', 'residual_sugar', 'sulphates', 'total_sulfur_dioxide',
'volatile_acidity']
CATEGORICAL_COLUMNS = ['quality']
feature_columns = []
for feature_name in NUMERIC_COLUMNS:
feature_columns.append(tf.feature_column.numeric_column(feature_name, dtype=tf.float32))
def make_input_fn(data_df, label_df, num_epochs=10, shuffle=True, batch_size=32):
def input_function():
ds = tf.data.Dataset.from_tensor_slices((dict(data_df), label_df))
if shuffle:
ds = ds.shuffle(1000)
ds = ds.batch(batch_size).repeat(num_epochs)
return ds
return input_function
train_input_fn = make_input_fn(x_train, y_train)
eval_input_fn = make_input_fn(x_test, y_test, num_epochs=1, shuffle=False)
linear_est = tf.estimator.LinearRegressor(
feature_columns=feature_columns,
model_dir=str(PATH) + "train"
)
linear_est.train(train_input_fn)
result = linear_est.evaluate(eval_input_fn)
print("--> OUTPUT = " + str(result))
def serving_input_receiver_fn():
inputs =
for feat in feature_columns:
inputs[feat.name] = tf.compat.v1.placeholder(shape=[None], dtype=feat.dtype)
print("--> INPUTS = " + str(inputs))
return tf.estimator.export.ServingInputReceiver(inputs, inputs)
linear_est.export_saved_model(export_dir_base=str(PATH) + "model", serving_input_receiver_fn=serving_input_receiver_fn)Enfin, nous créons un script python pour comparer l’efficacité du dernier modèle formé avec le modèle exécuté en production afin de toujours continuer à exécuter le meilleur modèle. Tout le code sera écrit dans un fichier nommé validation.py:
import pandas
import random
from pathlib import Path
import tensorflow as tf
import numpy as np
import sys
import os
PATH = "/mnt/data/"
model_dir = str(PATH) + "model"
subdirs = [x for x in Path(model_dir).iterdir()
if x.is_dir() and 'temp' not in str(x)]
latest = str(sorted(subdirs)[-1])
print("--> LATEST = " + str(latest))
model_prod_dir = str(PATH) + "model_prod"
if not os.path.exists(model_prod_dir):
os.makedirs(model_prod_dir)
subdirs_prod = [x for x in Path(model_prod_dir).iterdir()
if x.is_dir() and 'temp' not in str(x)]
if not subdirs_prod:
os.rename(latest, model_prod_dir + "/" + latest.split("/")[-1])
sys.exit(0)
latest_prod = str(sorted(subdirs_prod)[-1])
print("--> PROD = " + str(latest_prod))
randomlist = []
df = pandas.read_csv(str(PATH) + 'file.csv')
nb_raw = len(df)
for i in range(0, int((nb_raw/10))):
n = random.randint(0,nb_raw)
if n<nb_raw and n>=0:
randomlist.append(n)
else:
print(" _BAD_RANDOM_ ")
def build_predict(df, model):
res = model(chlorides=tf.constant(df['chlorides'], dtype=tf.float32, shape=1),
alcohol=tf.constant(df['alcohol'], dtype=tf.float32, shape=1),
citric_acid=tf.constant(df['citric_acid'], dtype=tf.float32, shape=1),
residual_sugar=tf.constant(df['residual_sugar'], dtype=tf.float32, shape=1),
total_sulfur_dioxide=tf.constant(df['total_sulfur_dioxide'], dtype=tf.float32, shape=1),
free_sulfur_dioxide=tf.constant(df['free_sulfur_dioxide'], dtype=tf.float32, shape=1),
pH=tf.constant(df['pH'], dtype=tf.float32, shape=1),
fixed_acidity=tf.constant(df['fixed_acidity'], dtype=tf.float32, shape=1),
sulphates=tf.constant(df['sulphates'], dtype=tf.float32, shape=1),
density=tf.constant(df['density'], dtype=tf.float32, shape=1),
volatile_acidity=tf.constant(df['volatile_acidity'], dtype=tf.float32, shape=1)
)
return res
model = tf.saved_model.load(export_dir=str(latest)).signatures['predict']
model_prod = tf.saved_model.load(export_dir=str(latest_prod)).signatures['predict']
pred = []
pred_prod = []
score_train=0
score_prod=0
for x in randomlist:
value = df.drop(["quality"], axis=1).iloc[x]
real = df['quality'].iloc[x]
pred_train = round(np.array(build_predict(value, model)['predictions'])[0][0])
if real == pred_train:
score_train += 1
pred_prod = round(np.array(build_predict(value, model_prod)['predictions'])[0][0])
if real == pred_prod:
score_prod += 1
print("score_train : " + str(score_train))
print("score_prod : " + str(score_prod))
if score_train > score_prod:
model_old_dir = str(PATH) + "model_old"
if not os.path.exists(model_old_dir):
os.makedirs(model_prod_dir)
os.rename(latest_prod, str(PATH) + "model_old/" + latest_prod.split("/")[-1])
os.rename(latest, model_prod_dir + "/" + latest.split("/")[-1])Création du pipeline
Nous allons maintenant créer un fichier YAML à la racine de notre répertoire de projet nommé liminal.yml. Déclarons d’abord nos volumes de montage. Pour cela, nous créons un volume Kubernetes nommé data lié au répertoire où notre liminal.yml fichier est localisé.
name: GettingStartedPipeline
volumes:
- volume: data
local:
path: .Ensuite, nous allons structurer et déclarer la commande de notre pipeline à l’aide d’un tasks. UN tasks est composé de plusieurs task et se caractérise par :
taskc’est le nom de la tâche (attention chaque tâche a un nom unique)typequi spécifie le type de scripts qui seront exécutés, dans notre cas, nous utilisons des scripts Pythondescriptionqui permet de décrire l’objectif de la tâcheimagequi spécifie à quelles images Docker le script sera associésourcequi indique le chemin où se trouve le scriptcmdqui permet d’aliaser la commande d’exécution du scriptmountsqui permet de monter le volume interne tel que défini ci-dessus dans un dossierenv_varsqui spécifie les variables d’environnement que nous voulons fournir à nos images.
Chaque tâche est exécutée par un DAG Airflow dans un pod distinct. Dans notre cas, ils partagent tous la même image Docker, déclarée dans le image champ, et le même volume spécifié dans le champ mounts champ.
name: GettingStartedPipeline
volumes:
- volume: data
local:
path: .
pipelines:
- pipeline: getting_started_pipeline
owner: Aargan
start_date: 1970-01-01
timeout_minutes: 10
schedule: 0 * 1 * *
default_array_loaded: [2, 3, 4]
default_object_loaded:
key1: val1
key2: val2
metrics:
namespace: TestNamespace
backends: [ ]
tasks:
- task: load_data
type: python
description: Load Dataset
image: python_hello_world_example_image
source: pythonscript
mounts:
- mount: mymount
volume: data
path: /mnt/data
cmd: python -u download.py
env_vars:
url: "https://raw.githubusercontent.com/mlflow/mlflow/master/examples/sklearn_elasticnet_wine/wine-quality.csv"
- task: training_model
type: python
description: training model
image: python_hello_world_example_image
source: pythonscript
mounts:
- mount: mymount
volume: data
path: /mnt/data
cmd: python -u wine_linear_regression.py
- task: validation_model
type: python
description: validation model
image: python_hello_world_example_image
source: pythonscript
mounts:
- mount: mymount
volume: data
path: /mnt/data
cmd: python -u validation.pyExécutez Apache Liminal
Maintenant, déployons notre pipeline à l’aide des commandes suivantes :
liminal build
liminal deploy --clean
liminal startApache Liminal est démarré. L’interface utilisateur d’Apache Airflow est accessible à l’adresse suivante : http://127.0.0.1:8080
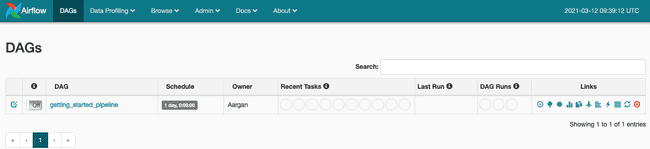
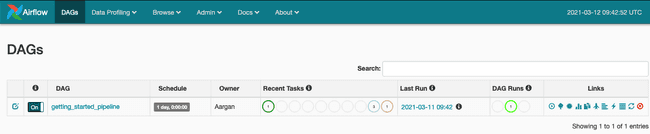
Activez simplement le DAG et le pipeline se déclenchera automatiquement.
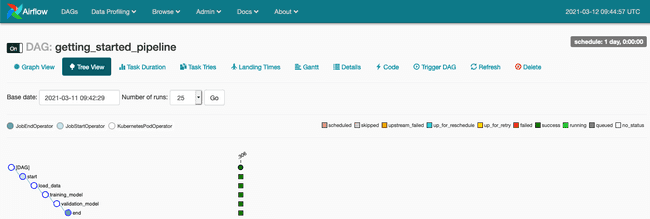
Nous suivons notre DAG et accédons aux journaux via le Tree View (voir notre article Présentation d’Apache Airflow sur AWS si vous souhaitez mieux comprendre les fonctionnalités d’Apache Airflow).
Une fois le pipeline entièrement exécuté et terminé, nous arrêtons notre serveur Liminal à l’aide de la commande :
Conclusion
Apache Liminal propose de simplifier la création de pipelines de Machine Learning de bout en bout. Nous pensons que l’initiative est un succès. En effet un seul fichier YAML vous permet de décrire de manière cohérente l’exécution de vos différents pipelines de Machine Learning.
De plus, l’utilisation de Kubernetes permet à l’utilisateur de déployer ses pipelines dans des clusters distants. Vous vous connectez à votre cluster distant à l’aide de la commande :
kubectl config set-context <your remote kubernetes cluster>Enfin l’utilisation de fichiers YAML déclaratifs présente l’avantage d’automatiser votre pipeline Machine Learning dans vos pipelines CI/CD afin de versionner, publier et exploiter vos modèles.